Barcoding gap
Praktikum na Barcoding gap a také na Species delimitation
Nejprve odkazy na stránky, které se budou hodit:
ABGD
home page https://bioinfo.mnhn.fr/abi/public/abgd/
užitečnější však bude rovnou https://bioinfo.mnhn.fr/abi/public/abgd/abgdweb.html
Notepad++ https://notepad-plus-plus.org/downloads/
FigTree https://github.com/rambaut/figtree/releases
MEGA https://www.megasoftware.net/
bPTP https://species.h-its.org/
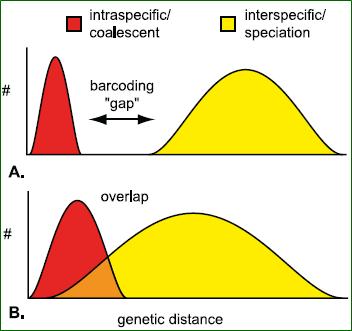
Barcoding gap je mezera mezi distribucí vnitro- a mezidruhových párových rozdílů. Koukněte na přednášku 2. Barcoding

Vnitrodruhové rozdíly by měly být menší než mezidruhové. (Není to ale vždy, viz přednáška.)
Tuto myšlenku využívají metody určení hranic druhů (species delimitation) pracující s genetickými vzdálenostmi. (Jsou ale taky metody, které pracují se stromy, třeba bPTP, viz odkaz nahoře, a spusta jiných.)
My jsme na praktiku zkoušeli vypočítat distance, ale nedotáhli jsme to do histogramů. Teď to napravíme.
Žádný strach. Úloha v základní podobě je nesmírně jednoduchá. Pro zájemce pak přidám i složitější věci na vyzkoušení.
Pozor! Cílem praktika je hlavně ukázat, že se toho člověk nemá děsit a jak zhruba vypadají programy. Pokud něco budete počítat do článku, tak je potřeba si to podrobně nastudovat a rozumět i nastavení těch programů!
Základní cvičení
Použijeme data z úlohy o homolicích. Připomínám, že jde o sekvence dvou druhů homolic Conus abbreviatus a Conus ebraeus.
Alignment je tu nebo v praktiku 2
Alignment si zkopírujte do schránky (normálka označit a CTRL-C)
Jdeme na stránku ABGD https://bioinfo.mnhn.fr/abi/public/abgd/abgdweb.html
Vidíme tu velké okno na data. No to už umíme, sem musíme zkopírovat naše data, tedy celý ten alignment (CTRL-V)
V nastavení zatím skoro vše necháme. Jen vedle zásadního čudlu "Go" si můžeme vybrat metodu výpočtu distancí.
Dáme třeba "simple". To jsou ty nejjednodušší p-distance, tedy procento rozdílů.
A teď už rychle to "Go".
Zjeví se stránka textu začínající /*
Co to říká? Inu zkusil to různě desetkrát (s různým apriorním limitem pro rozdělení do skupin) a vždy vyšlo, že to jsou dvě skupiny (2 groups), tedy asi dva druhy.
Klikněte na "here" a měly by se vám zjevit obrázky.
Vlevo je histogram. Na ose x jsou distance, na y jejich četnost. Vidíte krásnou mezeru mezi vnitro- a mezidruhovými distancemi?
Vpravo je v podstatě to samé. Mezera je ten velký skok v grafu.
Koukněte ale na graf dole. Není na první pohled úplně vzrušující, protože to vyšlo vždycky stejně.
Nicméně klikněte na nějaký bod v grafu (ty žluté puntíky). Zobrazí se vám, jak program rozhodil jedince do skupin. Zkontrolujte, zda jsou pohromadě jedinci stejného druhu (Názvy začínají Cab nebo Ceb jako C. abbreviatus a C. ebraeus) Stránku ještě nezavírejte, viz níže.
Toto je konec jednoduché úlohy. Nicméně zkuste aspoň první dvě z následujících doplňujících úloh. Je to zas celkem jednoduché a z výsledků můžete mít radost (budou obrázky!). Další dvě úlohy jsou docela dobré na to, abyste fakt porozuměli tomu, co činíte.
Doplňující cvičení 1
Zkusíme něco dalšího. Koukneme na strom konstruovaný z distancí pro dané rozdělení do skupin.
Na stránce s rozhozením jedinců do skupin zas dejte "here"
Zobrazí se strom v podobě závorkové konvence (text, závorky, čísla...). To by chtělo zobrazit jako strom, jinak je to děsný. Možností je spousta. Nejprve si strom zkopírujte a uložte jako textový soubor, třeba přes Notepad, tedy česky Poznámkový blok. Budete vypadat profesionálněji (fakt cool) pokud použijete Notepad ++, odkaz nahoře, ale pro tuto úlohu úplně stačí i obyčejný Notepad. Notepad ++ by se vám ale mohl hodit někdy příště :)
Zkusme třeba FigTree https://github.com/rambaut/figtree/releases
Po instalaci programu otevřeme náš soubor.
Chybí nám outgroup, tak to není moc pěkné. Pod slovem Layout jsou ale tři ikony. Zvolíme tu napravo. A ejhle, už máme pěkný nezakořeněný strom. Hezky se nám tu oddělili jedinci do dvou klastrů. Opět zkontrolujte, zda jsou pohromadě jedinci stejného druhu.
Komu se nelíbí FigTree, nechť zkusí třeba Dendroscope (stažení tu)
Doplňující cvičení 2
Zkuste vypočítat species delimitation přes bPTP https://species.h-its.org/ To je jeden z programů, co využívá rovnou stromy a ne jen distance.
Zadává se tam rovnou strom. Jeden strom (v té závorkové konvenci jako textový soubor) máme přeci z předchozí úlohy. Zkusíme ho tam nahrát (přes čudl Choose File).
Musíte tam zadat svůj e-mail (dole do okénka), jinak to nebude fungovat. V případě potřeby zkuste měnit nastavení. Na výsledky si chvíli počkáte. Zkuste se vyznat v tom, co to vlastně říká. Používá to dvě medoty - Maximum Likelihood a Bayes. Ukáže výsledky pro obě.
Znalce fylogenetických přístupů nepřekvapí "trace plot". Ten ukazuje, zda analýza běžela dostatečně dlouho a zda nemáme více odříznout "rozpačitý" začátek analýzy (Burn-in). Stručně: nevadí, když to nějak osciluje, vadí naopak jasný "trend". Trend znamená, že to furt leze nahoru nebo naopak dolu.
Doplňující cvičení 3
Zkuste tento soubor prohnat ABGD. Nebude to úplně jednoduché. Musíte jít do nastavení a trochu si s tím pohrát, konkrétně s "gap width".
Doplňující cvičení 4
Zkuste nejprve spočítat distance v programu MEGA (protože má daleko víc možností výpočtu distancí). MEGA se hodí, odkaz na stažení nahoře. Můžete zde zkusit i vybrat vhodný evoluční model pro výpočet distancí. Distance uložte jako soubor a ten pak prožeňte přes ABGD.